RDKitで記述子を計算
今回の記事ではRDKitを使って記述子を計算します。記述子計算をするときの注意点として、全ての記述子で同じ値になってしまうものがあるという点があげられます。このような記述子は削除する処理を行わないと、モデルにとってはただのノイズになってしまいます。以下では、記述子を計算し、その中から分散の小さい記述子を削除する流れをご説明いたします。
記述子の計算
次に、今回使用するパッケージをインポートします。記述子計算などにrdkit、図示にmatplotlibを使用しています。同時に、ヒストグラムを保存するディレクトリをグローバル変数として定義しておきます。
import os
import shutil
import pandas as pd
import matplotlib.pyplot as plt
from tqdm import tqdm
from rdkit import Chem
from rdkit.Chem import Descriptors
from rdkit.ML.Descriptors.MoleculeDescriptors import MolecularDescriptorCalculator
data_dir = "data"
histgram_dir = os.path.join(data_dir, "histgram")
low_std_image_dir = os.path.join(data_dir, "low_std")また、使用するデータは以前の記事で紹介したデータを使用します。そのままでは分かりにくいコードになってしまうので関数化したものを下に記載しています。
def read_csv(filename:str) -> pd.DataFrame:
"""CSVファイルの読み込み
Parameters
------
filename : str
CSVファイルパス
Returns
------
pd.DataFrame : SMILESと活性分類のデータフレーム
"""
# 2~6行目のヘッダーは不要なので読み込みをスキップ
# dtypeの指定はwarningを消すため
# DtypeWarning: Columns (0) have mixed types. Specify dtype option on import or set low_memory=False.
df = pd.read_csv(filename, skiprows=[1, 2, 3, 4, 5],
dtype={"PUBCHEM_RESULT_TAG":str})
# 最後の3行は必要データではないため削除
df = df.iloc[:-3]
# 必要な列の取り出し
df = df[["PUBCHEM_EXT_DATASOURCE_SMILES", "PUBCHEM_ACTIVITY_OUTCOME"]]
# 列名の変更
df.columns = ["SMILES", "ACTIVITY"]
# データがない行を削除
df.dropna(inplace=True)
df = df[df["SMILES"]!=""]
# Inactiveを0に、Activeを1に変換し、その他を削除する
df.loc[df["ACTIVITY"]=="Inactive", "ACTIVITY"] = 0
df.loc[df["ACTIVITY"]=="Active", "ACTIVITY"] = 1
df = df[(df["ACTIVITY"]==0) | (df["ACTIVITY"]==1)]
return dfそれでは次に記述子を計算するコードを記載します。
def calc_descriptor(csv_file:str, descriptor_file:str="descriptors.csv",
calc:bool=False) -> (pd.DataFrame, pd.DataFrame):
"""記述子を計算する
Parameters
------
csv_file : str
PUBCHEMからダウンロードしたCSVファイルパス
descriptor_file : str
記述子計算結果保存先/読み込み先のCSVファイルパス
calc : bool
記述子計算を行う. Falseなら保存している結果を読み込む.
"""
df = read_csv(csv_file)
# 活性分類を取得
y = df["ACTIVITY"]
if calc:
# SMILESからmolオブジェクトを取得
smiles = df["SMILES"]
mols = [Chem.MolFromSmiles(s) for s in tqdm(smiles)]
# 記述子計算
descriptors = [d[0] for d in Descriptors._descList]
descriptor_calculation = MolecularDescriptorCalculator(descriptors)
df_descriptor = pd.DataFrame(
[descriptor_calculation.CalcDescriptors(mol) for mol in tqdm(mols)],
columns=descriptors)
# 記述子計算に長時間かかるので、CSV保存
df_descriptor.to_csv(descriptor_file, index=None)
else:
df_descriptor = pd.read_csv(descriptor_file)
return df_descriptor, yread_csv関数を使ってpubchemからダウンロードしたCSVファイルを読み込みます。read_csv関数から返ってきたデータフレームはカラムとしてSMILESとACTIVTY(活性分類)を持っています。活性分類は今後モデルを作成する際に教師データとして使用するので変数yに値を格納します。
df = read_csv(csv_file)
# 活性分類を取得
y = df["ACTIVITY"]このデータフレームには24万行以上のデータが格納されています。従いまして、記述子計算には非常に時間がかかってしまいます。これを毎回繰り返していると作業がなかなか進まないので、一度計算した記述子はCSVファイルに保存しておき、次回以降はそのCSVファイルを読み込むことで記述子計算をスキップできるようにしておきます。
if calc:
# 中略
else:
df_descriptor = pd.read_csv(descriptor_file)記述子を計算するにはmolオブジェクトが必要になります。SMILESをmolオブジェクトに変換するにはrdkitのMolFromSmilesメソッドを使用します。また、tqdmライブラリを使用することでループの進捗状況をコンソールに出力することができます。
# SMILESからmolオブジェクトを取得
smiles = df["SMILES"]
mols = [Chem.MolFromSmiles(s) for s in tqdm(smiles)]続いてrdkitを使って記述子を計算します。計算可能な記述子の一覧はDescriptors._descListに格納されています。ここから一覧を取得し、descriptor_calculation.CalcDescriptorsメソッドにより記述子を計算します。最後にデータフレームをCSVファイルに保存すれば完了です。
# 記述子計算
descriptors = [d[0] for d in Descriptors._descList]
descriptor_calculation = MolecularDescriptorCalculator(descriptors)
df_descriptor = pd.DataFrame(
[descriptor_calculation.CalcDescriptors(mol) for mol in tqdm(mols)],
columns=descriptors)
# 記述子計算に長時間かかるので、CSV保存
df_descriptor.to_csv(descriptor_file, index=None)分散の小さい記述子の削除
ここからは分散の小さい記述子を削除していきます。最初に記述子削除用の関数であるdrop_low_var_col関数全体を掲載します。ここまでで解説してきたcalc_descriptor関数の1つ目の戻り値がdrop_low_var_col関数の一つ目の引数に入ることになります。
def drop_low_var_col(df:pd.DataFrame, thres_var:float=0.01, plot:bool=True) -> pd.DataFrame:
"""分散の少ない記述子を削除する.
Parameters
------
df : pd.DataFrame
記述子のデータフレーム
thres_var : float
削除する際の分散の閾値
plot : bool
記述子ごとのヒストグラムをプロットする
Returns
------
pd.DataFrame : 分散の少ない記述子を削除したデータフレーム
"""
# ヒストグラムを描画する時は保存先のディレクトリを作成する.
if plot:
os.makedirs(histgram_dir, exist_ok=True)
os.makedirs(low_std_image_dir, exist_ok=True)
low_std_col = []
# 記述子のデータフレームを1列ごとに処理
for (desc_name, col) in df.iteritems():
# 分散が閾値以下なら記述子名をリストに追加
if col.var() <= thres_var:
save_dir = low_std_image_dir
low_std_col.append(desc_name)
else:
save_dir = histgram_dir
if plot:
# ヒストグラムの作成、保存
col.hist()
plt.savefig(os.path.join(save_dir, f"{col.name}.png"))
plt.clf()
plt.close()
# 分散の少ない記述子を削除
df.drop(low_std_col, axis=1, inplace=True)
return df想定通り同じような値ばかりの記述子を削除できたか確かめるためにヒストグラムを作成しています。そのため最初にヒストグラム保存用のディレクトリを作成しています。このヒストグラム作成にも多少の時間がかかってしまうので、plot引数でヒストグラム作成のon, offができるようになっています。
# ヒストグラムを描画する時は保存先のディレクトリを作成する.
if plot:
os.makedirs(histgram_dir, exist_ok=True)
os.makedirs(low_std_image_dir, exist_ok=True)それぞれの記述子についてヒストグラムを作成したいということは、データフレームを1列ずつ処理したいということです。pandas.DataFrameを1列ずつ処理するにはiteritemsメソッドを使用します。これにより、desc_nameにはカラム名が、colにはpd.Series型でデータが格納されています。
pandasには、分散を計算するためのメソッドも準備されており、それがvarメソッドです。varメソッドにより計算した分散が、関数の引数で指定した閾値よりも小さければ、その記述子は削除されます。今回のデータでは、閾値として0.01を使用しています。
# 記述子のデータフレームを1列ごとに処理
for (desc_name, col) in df.iteritems():
# 分散が閾値以下なら記述子名をリストに追加
if col.var() <= thres_var:
save_dir = low_std_image_dir
low_std_col.append(desc_name)
else:
save_dir = histgram_dir列ごとのヒストグラムを作成します。pandasでは、histメソッドを使用することで簡単にヒストグラムを作成することができます。図を保存するときは記述子名がファイル名となるように保存します。
if plot:
# ヒストグラムの作成、保存
col.hist()
plt.savefig(os.path.join(save_dir, f"{col.name}.png"))
plt.clf()
plt.close()最後に分散が少なかった記述子の列をデータフレームから削除して終了です。
# 分散の少ない記述子を削除
df.drop(low_std_col, axis=1, inplace=True)コードの解説はここまでですが、分散の少なかった記述子のヒストグラムを確認しておきたいと思います。
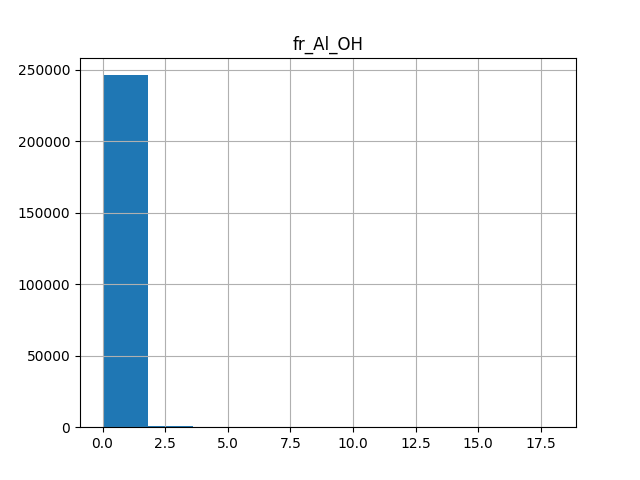
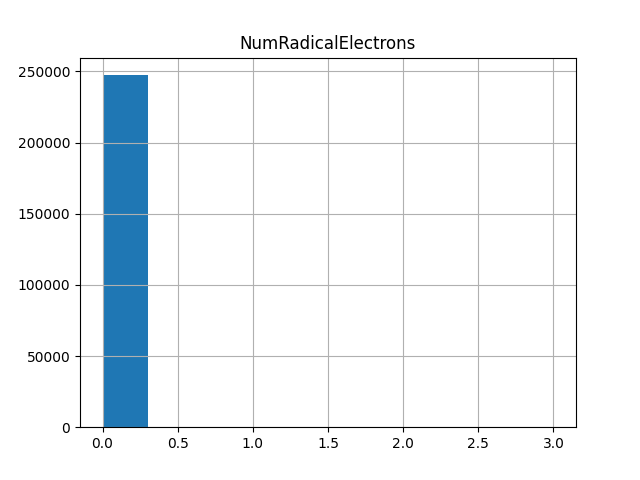
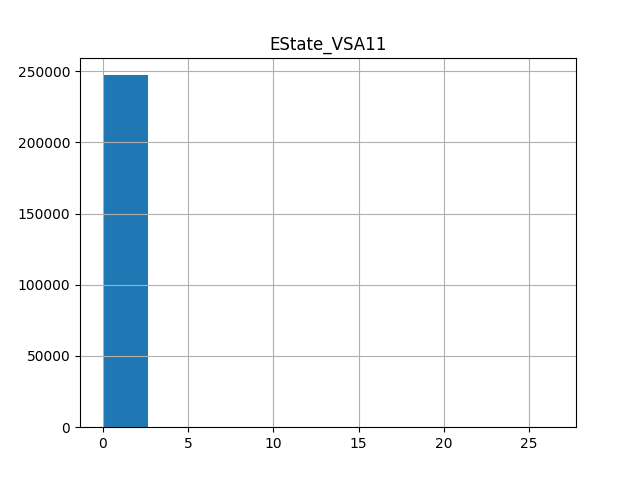
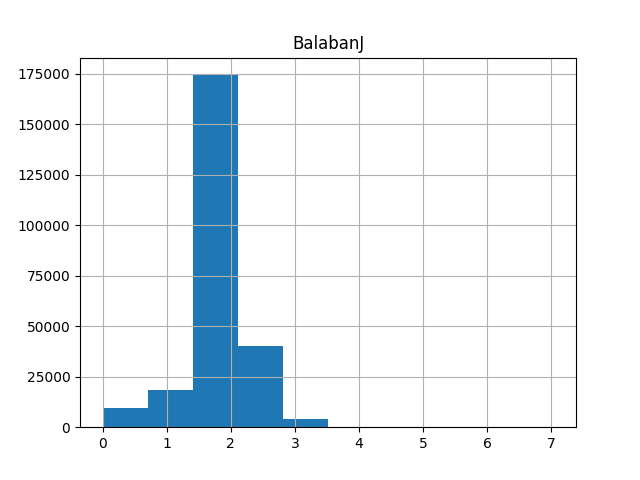
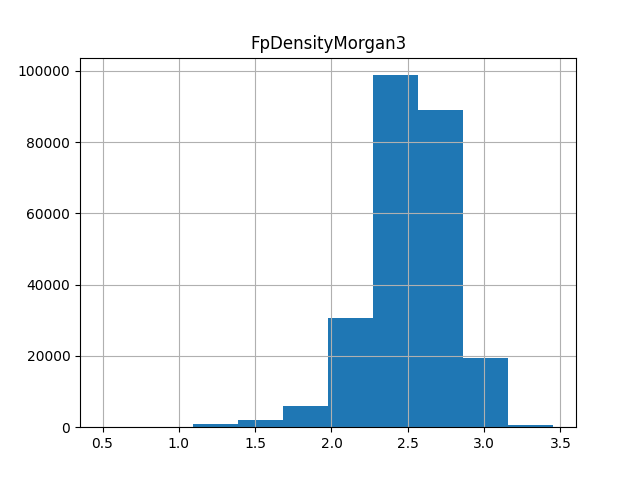
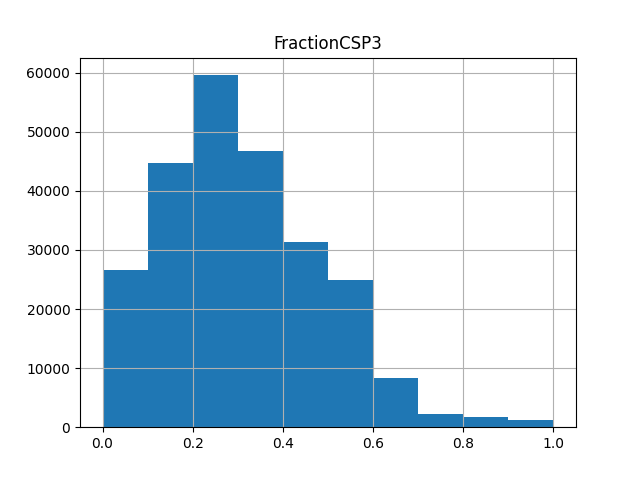
上側のグラフは削除していいグラフだと思いますが、下側のグラフは削除しない方がいいグラフだと思います。閾値の値についてはさらに検討の余地がありそうです。
最後に使用したコード全文を掲載して記事を終了したいと思います。ご覧いただきましてありがとうございました!
import os
import shutil
import pandas as pd
import matplotlib.pyplot as plt
from tqdm import tqdm
from rdkit import Chem
from rdkit.Chem import Descriptors
from rdkit.ML.Descriptors.MoleculeDescriptors import MolecularDescriptorCalculator
data_dir = "data"
histgram_dir = os.path.join(data_dir, "histgram")
low_std_image_dir = os.path.join(data_dir, "low_std")
def read_csv(filename:str) -> pd.DataFrame:
"""CSVファイルの読み込み
Parameters
------
filename : str
CSVファイルパス
Returns
------
pd.DataFrame : SMILESと活性分類のデータフレーム
"""
# 2~6行目のヘッダーは不要なので読み込みをスキップ
# dtypeの指定はwarningを消すため
# DtypeWarning: Columns (0) have mixed types. Specify dtype option on import or set low_memory=False.
df = pd.read_csv(filename, skiprows=[1, 2, 3, 4, 5],
dtype={"PUBCHEM_RESULT_TAG":str})
# 最後の3行は必要データではないため削除
df = df.iloc[:-3]
# 必要な列の取り出し
df = df[["PUBCHEM_EXT_DATASOURCE_SMILES", "PUBCHEM_ACTIVITY_OUTCOME"]]
# 列名の変更
df.columns = ["SMILES", "ACTIVITY"]
# データがない行を削除
df.dropna(inplace=True)
df = df[df["SMILES"]!=""]
# Inactiveを0に、Activeを1に変換し、その他を削除する
df.loc[df["ACTIVITY"]=="Inactive", "ACTIVITY"] = 0
df.loc[df["ACTIVITY"]=="Active", "ACTIVITY"] = 1
df = df[(df["ACTIVITY"]==0) | (df["ACTIVITY"]==1)]
return df
def calc_descriptor(csv_file:str, descriptor_file:str="descriptors.csv",
calc:bool=False) -> (pd.DataFrame, pd.DataFrame):
"""記述子を計算する
Parameters
------
csv_file : str
PUBCHEMからダウンロードしたCSVファイルパス
descriptor_file : str
記述子計算結果保存先/読み込み先のCSVファイルパス
calc : bool
記述子計算を行う. Falseなら保存している結果を読み込む.
"""
df = read_csv(csv_file)
# 活性分類を取得
y = df["ACTIVITY"]
# print(set(y))
if calc:
# SMILESからmolオブジェクトを取得
smiles = df["SMILES"]
mols = [Chem.MolFromSmiles(s) for s in tqdm(smiles)]
# 記述子計算
descriptors = [d[0] for d in Descriptors._descList]
descriptor_calculation = MolecularDescriptorCalculator(descriptors)
df_descriptor = pd.DataFrame(
[descriptor_calculation.CalcDescriptors(mol) for mol in tqdm(mols)],
columns=descriptors)
# 記述子計算に長時間かかるので、CSV保存
df_descriptor.to_csv(descriptor_file, index=None)
else:
df_descriptor = pd.read_csv(descriptor_file)
# print(df_descriptor)
return df_descriptor, y
def drop_low_var_col(df:pd.DataFrame, thres_var:float=0.01, plot:bool=True) -> pd.DataFrame:
"""分散の少ない記述子を削除する.
Parameters
------
df : pd.DataFrame
記述子のデータフレーム
thres_var : float
削除する際の分散の閾値
plot : bool
記述子ごとのヒストグラムをプロットする
Returns
------
pd.DataFrame : 分散の少ない記述子を削除したデータフレーム
"""
# ヒストグラムを描画する時は保存先のディレクトリを作成する.
if plot:
os.makedirs(histgram_dir, exist_ok=True)
os.makedirs(low_std_image_dir, exist_ok=True)
low_std_col = []
# 記述子のデータフレームを1列ごとに処理
for (desc_name, col) in df.iteritems():
# 分散が閾値以下なら記述子名をリストに追加
if col.var() <= thres_var:
save_dir = low_std_image_dir
low_std_col.append(desc_name)
else:
save_dir = histgram_dir
if plot:
# ヒストグラムの作成、保存
col.hist()
plt.title(col.name)
plt.savefig(os.path.join(save_dir, f"{col.name}.png"))
plt.clf()
plt.close()
# 分散の少ない記述子を削除
df.drop(low_std_col, axis=1, inplace=True)
return df
if __name__ == "__main__":
filename = "AID_504466_datatable_all.csv"
x, y = calc_descriptor(filename)
df = drop_low_var_col(x, True)
追記
– ライブラリのインストール方法
pip install pandas matplotlib tqdm rdkit-pypi