LightGBMの分類タスクでアンダーサンプリングとオーバーサンプリングの効果を検証する
この記事では、Pythonを用いた機械学習の手法の一つである、不均衡データの扱い方について説明します。不均衡データとは、データセットの中であるクラスのサンプル数が他のクラスに比べて圧倒的に少ない場合のことを指します。例えば、ある病気にかかる人の割合が1%しかいない場合や、ある商品を購入する人の割合が0.1%しかいない場合などが挙げられます。
不均衡データに対する機械学習の処理では、クラスのサンプル数が少ないため、モデルが正しく学習できずに偏った結果を出力することがあります。この問題に対処する方法の一つに、サンプル数の多いクラスから一部のデータを削除するundersamplingと、サンプル数の少ないクラスから新たにデータを作成するoversamplingがあります。
本記事では、pythonライブラリであるimbalanced-learnを用いてundersamplingのRandomUnderSamplerとoversamplingのSMOTEを用いたデータの前処理を行い、LightGBMを用いて分類モデルを作成する過程を示します。また、可視化の手法であるPCAを用いて、データ前処理後の分布を比較します。
環境構築
今回のコードを動かすために必要なPythonライブラリは、matplotlib, deepchem, imbalanced-learn, scikit-learn, lightgbmの5つです。これらはターミナルで以下のコマンドを実行することによりインストールすることができます。
pip install matplotlib deepchem imbalanced-learn scikit-learn lightgbmpipはPythonのパッケージ管理ツールで、パッケージをインストール・アップグレード・削除することができます。上記のコマンドでは、pipを使ってmatplotlib, deepchem, imbalanced-learn, scikit-learn, lightgbmを一括でインストールしています。ライブラリのインストールが完了すると、次のコードを実行することができます。
コード全文
import matplotlib.pyplot as plt
from deepchem.molnet import load_hiv
from imblearn.over_sampling import SMOTE
from imblearn.under_sampling import RandomUnderSampler
from sklearn.decomposition import PCA
from sklearn.metrics import accuracy_score, balanced_accuracy_score
import lightgbm as lgb
def run_pca(pca, x, y, filename, is_fit):
print(y[y == 0].shape, y[y == 1].shape)
if is_fit:
x_pca = pca.fit_transform(x)
else:
x_pca = pca.transform(x)
train_0 = x_pca[y == 0]
train_1 = x_pca[y == 1]
plt.scatter(train_0[:, 0], train_0[:, 1], label="label0")
plt.scatter(train_1[:, 0], train_1[:, 1], label="label1")
plt.legend()
plt.xlabel("PC1")
plt.ylabel("PC2")
plt.title("PC1 vs PC2")
plt.savefig(filename)
plt.clf()
plt.close()
tasks, datasets, transformers = load_hiv()
(train_dataset, valid_dataset, test_dataset) = datasets
x_train, y_train = train_dataset.X, train_dataset.y.ravel()
x_val, y_val = valid_dataset.X, valid_dataset.y.ravel()
x_test, y_test = test_dataset.X, test_dataset.y.ravel()
pca = PCA(n_components=2)
run_pca(pca, x_train, y_train, "original.png", True)
model = lgb.LGBMClassifier(max_depth=5, random_state=0)
model.fit(x_train, y_train)
pred = model.predict(x_val)
acc = accuracy_score(y_val, pred)
bacc = balanced_accuracy_score(y_val, pred)
print(acc, bacc)
rus = RandomUnderSampler(sampling_strategy=0.5, random_state=0)
x_train_resampled, y_train_resampled = rus.fit_resample(x_train, y_train)
run_pca(pca, x_train_resampled, y_train_resampled, "resample.png", False)
model = lgb.LGBMClassifier(max_depth=5, random_state=0)
model.fit(x_train_resampled, y_train_resampled)
pred = model.predict(x_val)
acc = accuracy_score(y_val, pred)
bacc = balanced_accuracy_score(y_val, pred)
print(acc, bacc)
smote = SMOTE(k_neighbors=5, random_state=0)
x_train_smote, y_train_smote = smote.fit_resample(x_train_resampled, y_train_resampled)
run_pca(pca, x_train_smote, y_train_smote, "smote.png", False)
model = lgb.LGBMClassifier(max_depth=5, random_state=0)
model.fit(x_train_smote, y_train_smote)
pred = model.predict(x_val)
acc = accuracy_score(y_val, pred)
bacc = balanced_accuracy_score(y_val, pred)
print(acc, bacc)コード解説
データセット
機械学習を行うには、適切なデータセットが必要です。しかし、データセットを用意することは容易ではありません。そこで、機械学習コミュニティでは、多くのデータセットが共有・提供されています。DeepChemは、機械学習のための化学データセットを提供するオープンソースのPythonライブラリです。DeepChemを使うことで、医薬品探索や材料設計など、化学に関する機械学習タスクを実行できます。
ここでは、DeepChemの機能を使って、HIVデータセットを生成しています。HIVデータセットは、実験的に測定された HIV の複製を阻害する活性を予測するために使用される分子のデータセットです。このデータセットには、HIV の複製を阻害する活性がある分子と活性がない分子の2クラスが含まれています。
tasks, datasets, transformers = load_hiv()
(train_dataset, valid_dataset, test_dataset) = datasets
x_train, y_train = train_dataset.X, train_dataset.y.ravel()
x_val, y_val = valid_dataset.X, valid_dataset.y.ravel()
x_test, y_test = test_dataset.X, test_dataset.y.ravel()上記のコードでは、load_hiv()関数を使用してHIVデータセットを取得しています。この関数は、3つのオブジェクトを返します。1つはタスクを示すオブジェクト、もう1つはtrain、validation、testのデータを含むオブジェクト、そして最後にトランスフォーマーのオブジェクトがあります。今回は各データを含むオブジェクトのみを使用します。各データはそれぞれ説明変数と目的変数に分かれています。コード上ではそれらをx, yとして表現しています。
データの可視化
データの可視化はrun_pca関数で行っています。run_pca関数は、PCA(Principal Component Analysis)というデータ分析手法を利用して、2次元の散布図を生成するための関数です。
def run_pca(pca, x, y, filename, is_fit):
print(y[y == 0].shape, y[y == 1].shape)
if is_fit:
x_pca = pca.fit_transform(x)
else:
x_pca = pca.transform(x)
train_0 = x_pca[y == 0]
train_1 = x_pca[y == 1]
plt.scatter(train_0[:, 0], train_0[:, 1], label="label0")
plt.scatter(train_1[:, 0], train_1[:, 1], label="label1")
plt.legend()
plt.xlabel("PC1")
plt.ylabel("PC2")
plt.title("PC1 vs PC2")
plt.savefig(filename)
plt.clf()
plt.close()この関数は、以下のように引数を受け取ります。
- pca: PCAオブジェクト。PCAを実行するために使用されます。
- x: PCAを適用するデータの特徴量。numpy配列で表現されます。
- y: PCAを適用するデータのラベル。numpy配列で表現されます。
- filename: 生成する散布図の画像ファイルの名前。
- is_fit: PCAの学習を実行するかどうかを指定するフラグ。
下の図は、HIVデータセットのtrainデータに対してPCAを行った結果です。
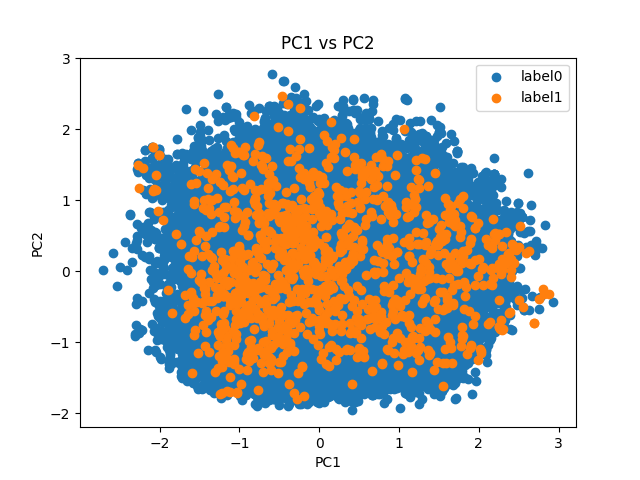
PCAの結果からも活性ありのラベルが極端に少ないことがわかります。実際の数値は活性なしが31669個、活性ありが1232個です。
分類精度の算出
データの可視化を行った後は、LightGBMを訓練し、validationデータセットで予測を行い、精度評価を行っています。
model = lgb.LGBMClassifier(max_depth=5, random_state=0)
model.fit(x_train, y_train)
pred = model.predict(x_val)
acc = accuracy_score(y_val, pred)
bacc = balanced_accuracy_score(y_val, pred)
print(acc, bacc)LGBMClassifierを使用して、深さ5の決定木で構成されるLightGBMの分類器オブジェクトを生成し、fit関数を使用して訓練データセットに対してモデルを訓練します。訓練が完了したら、predict関数を使用してvalidationデータセットに対して予測を行い、accuracyとbalanced accuracyにより精度評価をしています。accuracyは正解率で、balanced accuracyはサンプル数の偏りを考慮した正解率で、ラベル事の正解率の平均となります。今回は不均衡データを用いて予測を行うのでaccuracyだけではなくbalanced accuracyも用いて評価を行っています。
validationデータに対するaccuracyとbalanced accuracyはそれぞれ0.982と0.561となりました。確認はしていませんが、恐らく大量に存在する非活性サンプルの分類はうまく行うことができているためaccuracyは0.982と大きい値となり、逆に活性サンプルの分類がうまく行えていないためblanced accuracyは0.561しか出ていないと考えられます。活性サンプルをうまく分類することがこのモデルの課題となりそうです。
アンダーサンプリング
rus = RandomUnderSampler(sampling_strategy=0.5, random_state=0)
x_train_resampled, y_train_resampled = rus.fit_resample(x_train, y_train)不均衡データの場合、モデルは多数派クラスに偏って学習してしまい、少数派クラスの予測精度が低下する傾向があります。Random Under Samplingは、多数派クラスからランダムにサンプルを削除することでデータセットのバランスを改善する手法の一つです。上記のコードでは、sampling_strategyでサンプリング後の少数派クラスの割合を設定しています。0.5を指定しているため、少数派クラスが多数派クラスの50%になるようにサンプリングされています。RandomUnderSamplerはimbalanced-learnライブラリの中に実装されており、上記のコードのように簡単に実行できます。実行すると、少数派クラスのサンプル数が減少し、データセットがバランスがとれた状態になります。
アンダーサンプリングにより非活性サンプルが2464個、活性サンプルが1232個となりました。このデータセットに対してPCAを行った結果は以下のようになります。

元のデータセットに対してPCAを行ったものと比べると青色で示される非活性サンプルの数が明らかに少なくなっていることがわかります。
さらにLightGBMを使って精度評価を行うと、accuracy=0.961, balanced_accuracy=0.678となりました。accuracyは下がってしまいましたが、balanced accuracyの値は0.117も大きくなっています。これはサンプル数の少なかった活性サンプルの分類がうまくできていることが要因だと考えられます。
オーバーサンプリング
今回のコードでは、不均衡データの問題を解決するためにアンダーサンプリングと同時に、SMOTE (Synthetic Minority Over-sampling Technique) を用いてオーバーサンプリングを行っています。SMOTEはK近傍法を使っており、近傍の少数派サンプルから新しい少数派サンプルを生み出しています。
smote = SMOTE(k_neighbors=5, random_state=0)
x_train_smote, y_train_smote = smote.fit_resample(x_train_resampled, y_train_resampled)k_neighbors引数はK近傍法を使う際の近傍点の数を表すパラメータです。imbalanced-learnライブラリを使うとオーバーサンプリングについても上記のように簡単に実装することができます。
アンダーサンプリングにより非活性サンプルが2464個、活性サンプルが2464個となりました。このデータセットに対してPCAを行った結果は以下のようになります。

活性サンプルを表すオレンジ色の点が、今までのものと比べて多くなっていることがわかります。
このデータセットに対してもLightGBMを用いて精度評価を行いました。その結果は、accuracy=0.942, balanced_accuracy=0.674となりました。アンダーサンプリングのみの時と比べて精度は落ちてしまいましたが、もとのデータセットに比べるとbalanced accuracyの値が大きくなっていることがわかります。
まとめ
今回はHIV阻害活性についての構造活性相関のタスクにおいて、不均衡データに対する前処理を行うことにより予測精度を向上させる手法をご紹介しました。
不均衡データに対する前処理をまとめます。
- アンダーサンプリング:多数派クラスのデータを削除することで多数派クラスのデータ数を減らす手法
- オーバーサンプリング:少数派クラスのデータを新しく生み出してデータ数を増やす手法
弊社では、こうした高度なデータ解析技術を駆使して、お客様の課題を解決するためのソリューションを提供しています。データの前処理からモデル構築、運用まで、一貫してサポートすることで、お客様のビジネス価値を最大化することができます。是非、お気軽にご相談ください。