PubChem bioassayデータをpandas.DataFrameで処理する
PubChemは無料で利用できる世界最大級の化学分子データベースです。今回はPubChemで取得できるbioassayデータをpythonのpandasライブラリを用いて処理する方法を紹介します。
bioassayデータをダウンロード
最初にPubChemからbioassayデータをダウンロードします。今回はPubChem AID 504466を使用することにしたいと思います。ブラウザで「PubChem AID 504466」と検索すると以下のページを見つけることができます。
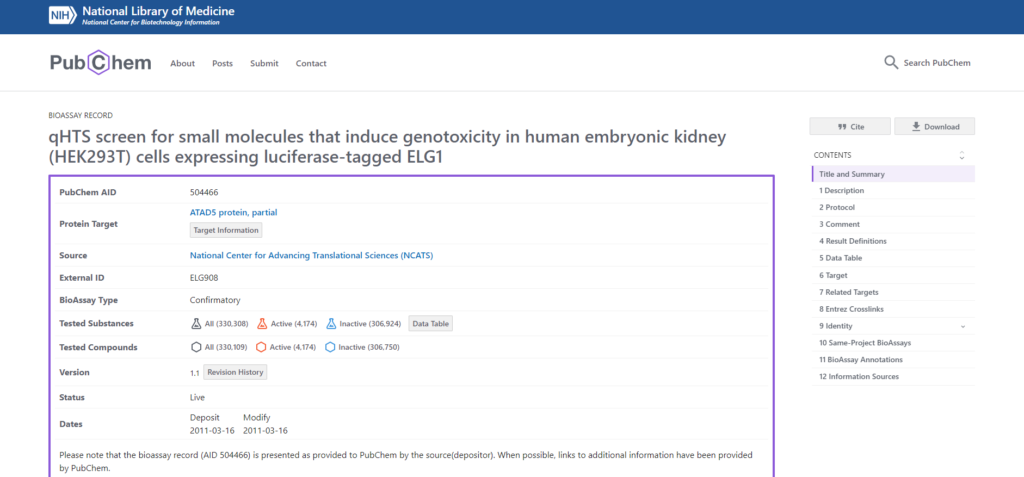
下にスクロールしていくと5 Data Tableという項目があり、右側にDownloadのボタンがあります。
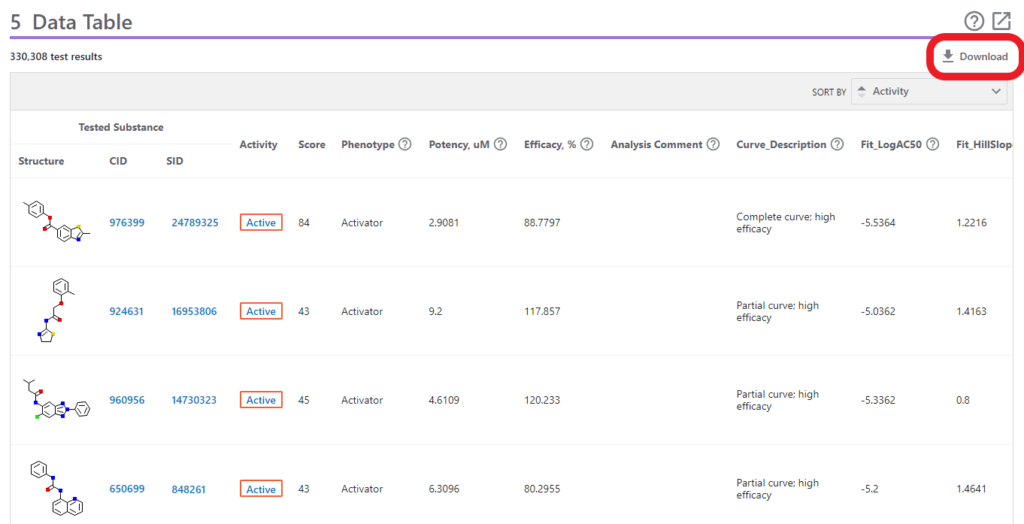
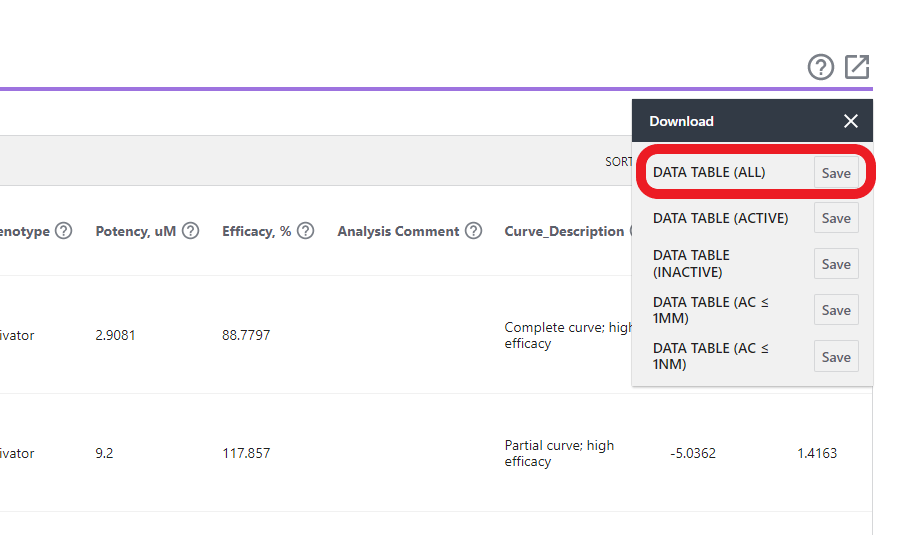
Downloadをクリックするといくつか選択肢が出てくるので、DATA TABLE (ALL)のSaveをクリックするとダウンロードが始まります。
pandasのインストール
Pythonを使ったCSVの取り扱いにはpandasを利用すると便利です。pandasはpipでインストール可能です。
pip install pandaspandas.DataFrameでのCSV読み込み
最初にpandasを使ったCSVの読み込みのコードを記載します。
import pandas as pd
# ファイル名
filename = "AID_504466_datatable_all.csv"
# 2~6行目のヘッダーは不要なので読み込みをスキップ
df = pd.read_csv(filename, skiprows=[1, 2, 3, 4, 5])
# 必要な列の取り出し
df = df[["PUBCHEM_EXT_DATASOURCE_SMILES", "PUBCHEM_ACTIVITY_OUTCOME"]]
# 列名の変更
df.columns = ["SMILES", "ACTIVITY"]
# データがない行を削除
df.dropna(inplace=True)
df = df[df["SMILES"]!=""]
# Inactiveを0に、Activeを1に変換し、その他を削除する。
df.loc[df["ACTIVITY"]=="Inactive", "ACTIVITY"] = 0
df.loc[df["ACTIVITY"]=="Active", "ACTIVITY"] = 1
df = df[(df["ACTIVITY"]==0) | (df["ACTIVITY"]==1)]ダウンロードしたファイルはAID_504466_datatable_all.csvというファイル名になっています。これをpythonファイルと同じ階層に置きます。
pandasでCSVファイルを読み込む時はread_csvを使用します。今回のCSVファイルはヘッダー行が複数ありますが、処理の際に必要になりそうなのは1行目だけです。そこでskiprows引数で不要なヘッダー行の読み込みをスキップしています。
PubChemのCSVはここまでpandas.DataFrameとして読み込むことができました。この後は簡単なデータ処理を行っています。
例えば、SMILESとACTIVITY_OUTCOMEだけ必要であれば、上記スクリプトのように列指定して2列だけ抽出することが可能です。もともとの列名は少し長いので短くてわかりやすい名前に変更しています。
CSVに値がなかった場合、pandas.DataFrameではnanが入っています。dropnaを利用することでnanが存在する行を削除することができます。inplece=Trueでdf変数に格納されているデータを変更します。また、SMILES列には空文字列があることがありますので、空文字列は削除しています。
その後、ACTIVITY列を数値に変換します。今後、InactiveとActiveの分類を行おうと考えているので、Inactiveを0にActiveを1に変換し、それ以外のデータを削除します。
以上のような流れで、PubChemのbioassayデータをpythonのpandas.DataFrameで処理することができます。